核磁共振是鉴定有机化台物结最重要的方法,但解析复杂核磁谱却愁坏了不少化学同仁,许多小伙伴(高手除外)直呼:解谱太难啦!
不久前,英国剑桥大学Goodman等人开发了一个电脑程序DP4-AI,该程序可以自动解析1H和13C NMR数据,分配峰,并给出一组最有可能的结构。DP4-AI“为结构不确定性提供了全自动解决方案,节省了解析NMR光谱的时间,同时也使分析充满信心。”
在有机合成和天然产物化学中,分子结构的确证都是具有挑战性的问题。结构上接近的异构体(例如区域异构体)和非对映异构体通常在1D-NMR光谱中仅表现出细微的差异,因此很难确定其结构和相对立体化学。虽然可以通过其他NMR实验(例如NOE光谱或合成天然产物的异构体),然后将所得观察到的NMR光谱与公开的NMR光谱进行比较,来解决这一问题,但两种方法都非常昂贵且耗时。
多年来,自动解析NMR光谱一直是结构化学的主要目标。DP4程序应运而生,成了一种非常具有吸引力的方法,DP4不仅可以预测分子的相对立体化学和其他变化,而且可以使用贝叶斯定理给出每个候选分子是正确分子的可能性,现已成功用于确定许多天然产物的立体化学。自发布以来,DP4的计算已得到简化,并且用户输入最小化,所有计算现在都由Python程序PyDP4自动管理。
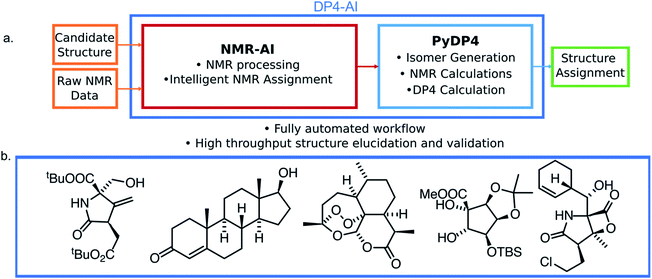
图1 (a)DP4-AI的结构。该系统提供了全自动的立体化学说明,用户只需输入原始NMR数据。(b)具有立体化学的示例结构可以使用集成在PyDP4中的DP4-AI完全自动地正确预测。
标准DP4要求用户输入带有立体化学不确定的分子结构和1D 13 C和1 H NMR光谱,其中最耗费精力的部分是峰的分配,而且容易出错。DP4-AI的目的是消除这种人工输入。它可以获取原始NMR数据并自动处理,分析光谱并将其与DFT计算的光谱进行比较,以输出对试验结构的置信度的定量度量。与手动过程相比,DP4-AI可以在大约60秒内完成一个分子的完整计算,而这可能需要人工花费8个小时的时间。这与商业软件Mnova不同。Mnova旨在帮助用户处理和解释其光谱,而DP4-AI使用分配例程结合DFT计算。
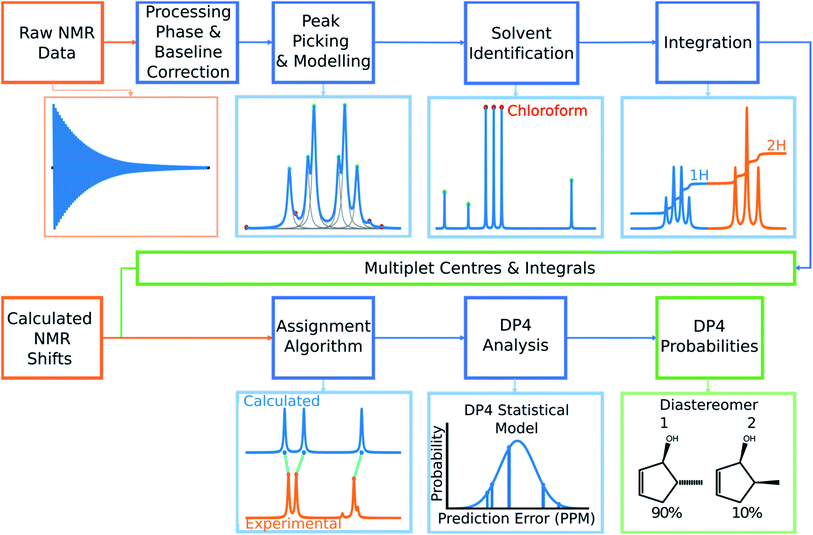
图2 DP4-AI在一系列阶段中处理原始NMR数据,以产生实验的多重位移值及其积分。然后,程序对分子中的每个原子进行使用DFT计算的位移,并将其分配给实验峰。然后使用该分配为每个非对映异构体计算DP4概率
研究人员在47个分子上评估了DP4-AI,每个分子平均具有3.49立体中心和不同范围的碳骨架。令人印象深刻的是,DP4-AI在32和64个非对映异构体中正确分配了分子NP1和NP2的相对立体化学。
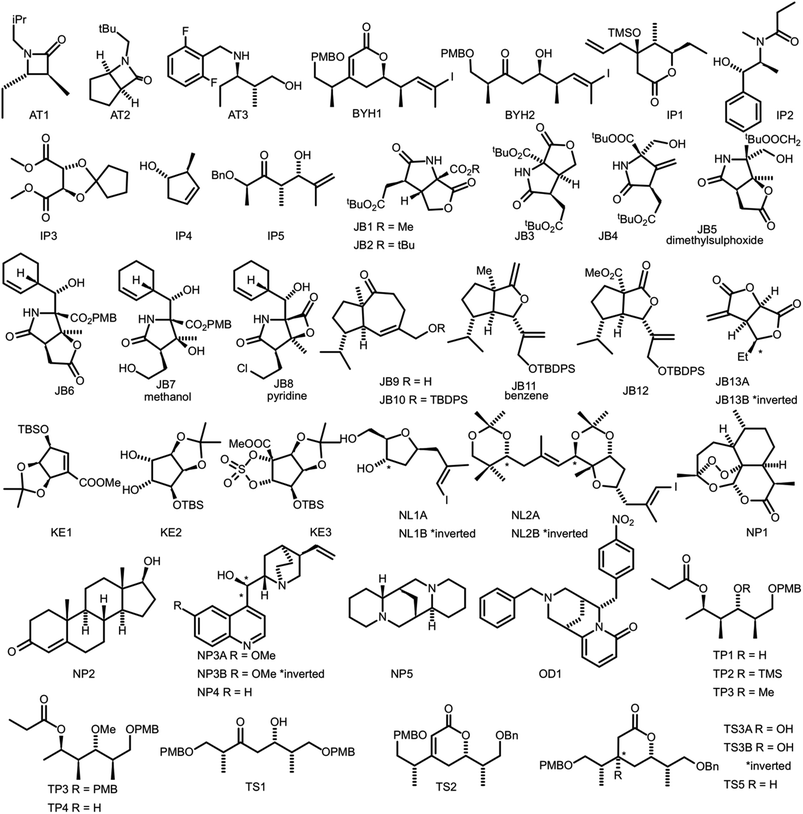
DP4-AI 作为一种强大的系统,可通过自动处理和分配原始13 C和1 H NMR光谱自动解决结构不确定性,并已作为开源软件发布。这种自动化将允许对数据库和大量分子进行快速DP4分析,这在以前是不可能的。利用由专业化学家撰写的NMR描述,DP4-AI保持与DP4相同的正确率。此外,该系统可以可靠地处理和分配NMR谱,速度约快60倍,节省了大量科研人员的时间。此外,该新系统为将来开发新功能(例如J值分析,2D-NMR分配,分配复杂混合物的光谱和辅助构象分析)提供了强大的框架。
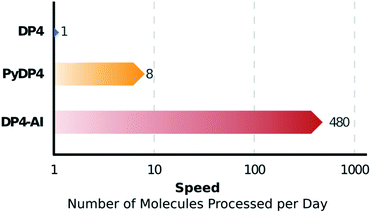
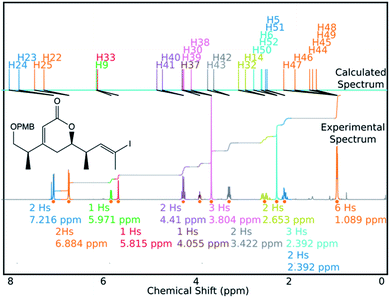
图5 NMR-AI可以在一分钟左右的时间内完成一个分子用于DP4的计算,这一任务以前需要大约8个小时的用户时间。这相当于每天可处理的分子数量增加约60倍。
Goodman并不担心自动化过程的发展会降低有机化学家手动分析NMR光谱的能力。计算器并没有阻止人们进行算术,而是允许人们更快,更准确地执行复杂的算术。他计划探索如何将DP4-AI扩展到其他原子核,而不仅仅是1 H和13C。
“我们对化学界对DP4-A的想法感到兴奋。任何人都可以以自己的方式使用和编辑系统来解决有趣的问题(人们已经是)。我们将非常感兴趣地看到人们将来如何使用和开发该软件。”
DP4-AI可作为开源软件在https://github.com/KristapsE/DP4-AI处获得。
开源软件:http://www-jmg.ch.cam.ac.uk/tools/nmr/
https://github.com/KristapsE/DP4-AI
需Python环境:https://www.python.org/downloads/
参考文献
A Howarth,K Ermanis和JM Goodman,Chemical Science,2020,DOI:10.1039 / d0sc00442a
https://pubs.rsc.org/en/content/articlehtml/2020/sc/d0sc00442a




